|
| | Esta página contém maiores detalhes sobre o software
Phred.

 | Objetivos
O principal objetivo do Phred é realizar a transformação de cromatogramas
(traces), provenientes das máquinas de seqüenciamento, em seqüências de
DNA. Baseado na análise do cromatograma, ele também associa um fator de
qualidade para cada base da seqüência gerada. Esse fator, como será
explicado a seguir, é de enorme valor para as demais fases do
seqüenciamento de DNA.
|
 |
Interface
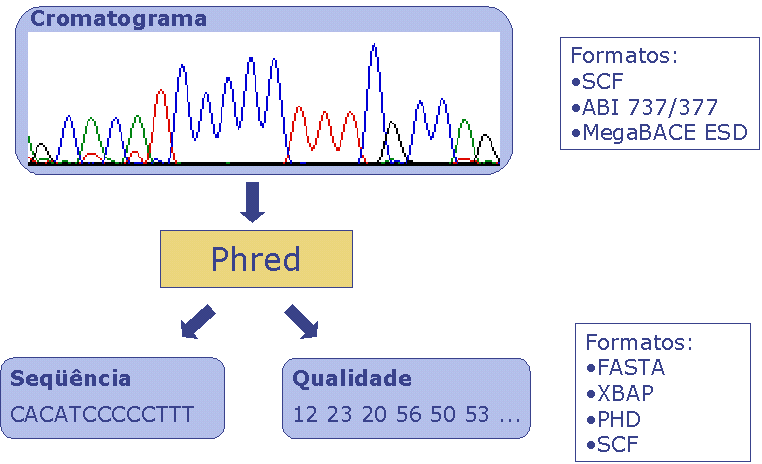
A seguir é ilustrada a comunicação com o software Phred. Basicamente
ela se dá via arquivos, que podem ter diversos formatos, tanto de entrada
quanto de saída. A figura ilustra estes formatos:
|
 |
Fator de Qualidade
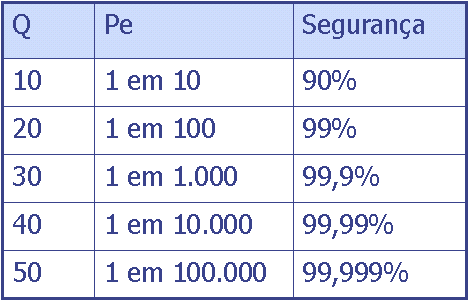
Um dos pontos fortes do Phred é a geração de arquivos de qualidade
descrevendo a confiança que pode-se ter em cada base nomeada. A qualidade
de cada base varia entre 4 e 60 e indica a chance da base estar correta.
Dessa forma, quanto maior a qualidade melhor.
O valor da qualidade é
determinado pela análise do cromatograma na vizinhança de cada base. A
seguir é mostrada uma tabela com a relação entre o valor de qualidade (Q)
e o nível de confiança que pode-se assumir. Também é mostrada a fórmula
utilizada pelo Phed para a geração desses valores.
|
 |
Exemplo
Como ilustração de uso do Phred, é mostrado a seguir o resultado da
execução do Phred sobre o arquivo de cromatograma (no formato ABI 377) LCP5AGGEU!LIKAA05.g.
A execução produz dois arquivos no formato FASTA. Um contendo a
seqüência nomeada e outro contendo os valores de qualidade para cada base
na seqüência.

|
 | Visualização usando TraceViewer
Como foi mencionado, o Phred comunica-se via arquivos, ou seja, ele não possui recursos de visualização do
cromatograma, o que torna bastante difícil a análise da qualidade do seu resultado.
Para prover esse recurso, alguns softwares já foram desenvolvidos com o
objetivo de mostrar o trace de entrada para o Phred (um cromatograma), as
decisões tomadas pelo Phred (bases nomeadas) e a qualidade de cada base.
Entre esse programas, um que merece destaque é o TraceViewer.
Primeiro porque ele permite o upload de cromatogramas para um servidor
potente, onde será executado o Phred sobre ele, segundo porque ele permite
a visualização do resultado em um browser através de um applet simples,
mas muito bom.
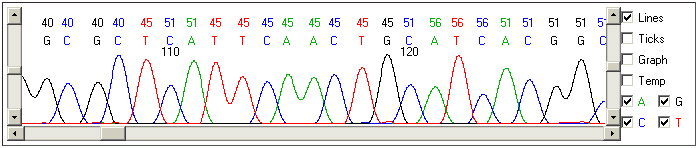
Abaixo estão dois exemplos de trechos do arquivo de cromatograma mencionado
acima.
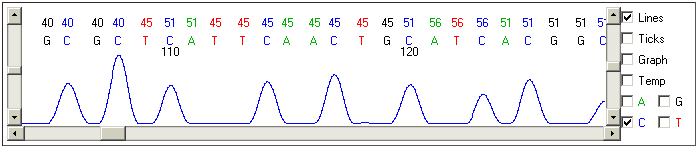
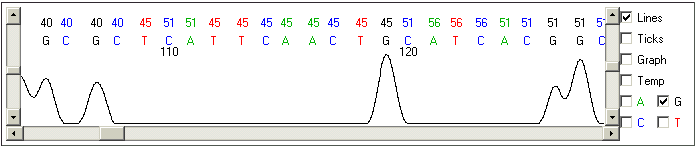
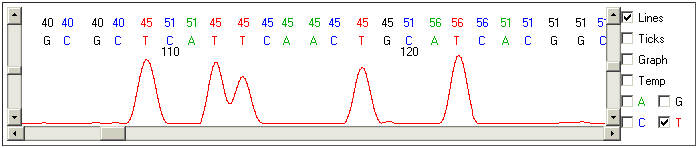
Uma parte de excelente qualidade (bases de 105-128). Observar que os
gráficos de cada tipo de base (ACGT) se expressam claramente e apenas nos pontos
em que eles irão nomear bases daquele tipo. Ou seja, eles contribuem onde
é necessário e não atrapalham em outros pontos.
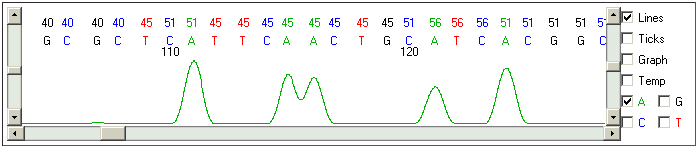
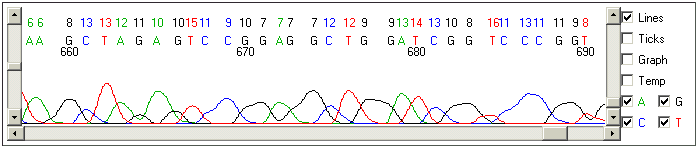
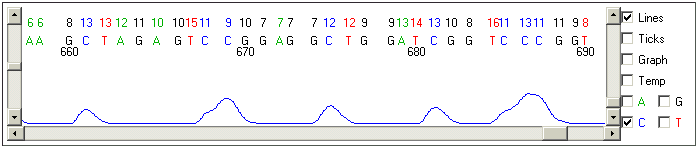
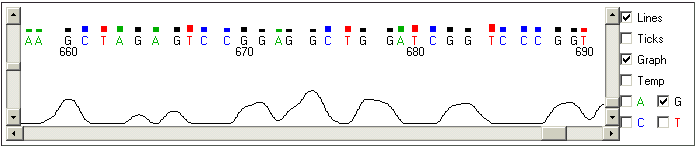
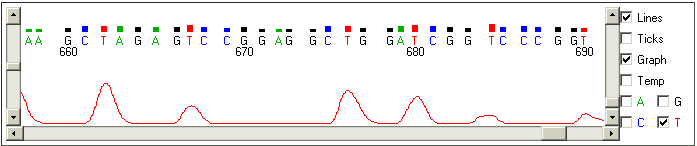
Uma parte de qualidade muito inferior (bases de 658-690). Observar que os
gráficos de cada tipo de base se expressam de forma muito inferior e não
apenas nos pontos em que eles irão nomear bases daquele tipo. Ou seja, eles
atrapalham e produzem ambigüidades em certos pontos, levando a baixos
valores de qualidade.

|
 |
Parâmetros de execução
Ao executar o Phred, 50 parâmetros podem ser especificados de acordo com a
necessidade do usuário. Por exemplo, diretório dos arquivos de entrada/saída,
tipo do formato de saída, rejeitar bases cuja qualidade seja inferior a um
limite especificado, etc.
|
 | Importância dos fatores de qualidade
Como foi observado, a importância dos arquivos de qualidade é muito
grande. Para explicitar essa importância, cosidere o cenário onde não há
tais arquivos. Quando isso ocorre, usualmente é atribuído um fator de
qualidade padrão para todas as bases. Dessa forma, corre-se o risco de
prejudicar toda a análise da seqüência, pois trechos bons irão receber
fatores abaixo do normal, enquanto que trechos ruins recebem valores
melhores que o esperado.
Dessa forma, é extremamente recomendável utilizar os fatores de qualidade
durante as demais fases do processo de seqüenciamento. O único cenário em
que tais valores não são aplicáveis é quando não tem-se acesso aos
arquivos de cromatogramas, apenas às seqüências já em formato FASTA.
|
|